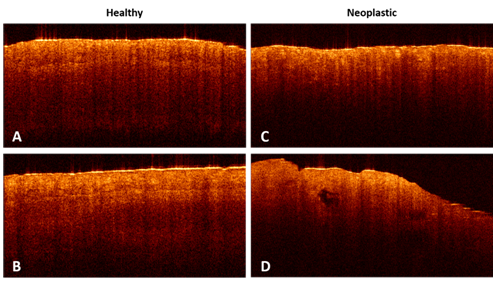
Figura: Ejemplo de imágenes OCT coloreadas. Izquierda: tejido sano. Derecha: pólipo neoplásico.
Versión preliminar (v1)
* Descripción general:
Esta base de datos consiste en una colección de imágenes de Tomografía de Coherencia Óptica (OCT) adquiridas con un sistema comercial de configuración spectral domain (Thorlbas Callisto) a partes de pólipos de colon y tejido sano provenientes de muestras de modelo animal.
Las muestras se han extraído a partir de un grupo de 60 ratas neoplásicas (del modelo clínicamente validad Pirc F344/NTac-Apcam1137) y 30 ratas hiperplásicas (creadas a partir de un modelo de hiperplasia inducida). Las muestras sanas se han obtenido de un grupo de 10 animales de control o de tejido sin lesión del grupo de ratas hiperplásicas.
La base de datos está formada por los siguientes ejemplos:
-tejido sano (10 muestras, 48 C-scans, 12820 B-scans)
-pólipo hiperplásico (13 muestras, 53 C-scans, 13903 B-scans)
-pólipo neoplásico (75 muestras, 245 C-scans, 67964 B-scans)
*C-scan: volumen 3D formado a partir de imágenes B-scan consecutivas
* Detalles técnicos:
La base de datos se organiza en carpetas en función del diagnóstico objetivo como se describe anteriormente. Después, la carpeta de cada muestra está etiquetada de la siguiente manera:
ORIGEN_ID_ANIMAL_ID_SECCION_N_MUESTRAS_SECCION_LESION_ID_NUM_LESIONES_SECUENCIA, donde:
ORIGEN_ID: Grupo al que pertenece el espécimen
ANIMAL_ID: Identificador del espécimen dentro del grupo
SECCION: Sección del colon donde se localiza la muestra (A-ascendente, T-transverso, D-descendente)
N_MUESTRAS_SECCION: Numero de muestras analizadas en la sección actual
LESION_ID: Identificador de la lesión analizada
NUM_LESIONES: Número total de lesiones de la muestra
SECUENCIA: Secuencia identificadora del C-scan. Debido al tamaño de los archivos, las lesiones se pueden dividir en varias carpetas.
Para acceder a las imágenes es necesario rellenar el siguiente formulario: https://forms.office.com/r/E8Aw9bpv4H o contactar con el Biobanco Vasco en solicitudes.biobancovasco@bioef.eus donde se informará de las condiciones de acceso.
Por favor, cita la siguiente referencia cuando uses esta base de datos:
Saratxaga, C.L.; Bote, J.; Ortega-Morán, J.F.; Picón, A.; Terradillos, E.; del Río, N.A.; Andraka, N.; Garrote, E.; Conde, O.M. Characterization of Optical Coherence Tomography Images for Colon Lesion Differentiation under Deep Learning. Appl. Sci. 2021, 11, 3119. https://doi.org/10.3390/app11073119.
Versión definitiva (v2)
NOTA: Esta base de datos estará disponible cuando se publique el artículo científico asociado.
El uso de la base de datos está restringido para fines educativos y de investigación. El uso con fines comerciales está prohibido sin autorización previa por escrita.